近日,動物科技學(xué)院張勤教授團隊在奶牛行業(yè)國際著名期刊《Journal of Dairy Science》在線發(fā)表了題為“Longitudinal genome-wide association studies of milk production traits in Holstein cattle using whole-genome sequence data imputed from medium-density chip data”的研究論文。團隊博士后滕軍和王丹教授為該論文的第一作者,張勤教授和寧超副教授為該論文的通訊作者。
縱向性狀(例如奶牛的產(chǎn)奶性狀)指的是一個個體有多個表型測量值,并且表型值隨時間發(fā)生動態(tài)變化。每個時間點的測量值稱為測定日記錄,使用測定日記錄分析縱向性狀可以最大限度地減少環(huán)境噪聲,從而提高檢驗功效。以往奶牛產(chǎn)奶性狀的全基因組關(guān)聯(lián)分析研究,要么基于測定日記錄(隨機回歸測定日模型),但是使用的芯片數(shù)據(jù);要么使用填充的序列數(shù)據(jù),但是基于偽表型(例如女兒產(chǎn)量離差或逆回歸育種值;傳統(tǒng)動物模型)。
該研究以千牛基因組計劃中1059頭荷斯坦牛基因型為參考面板,將6470頭中國荷斯坦牛的芯片數(shù)據(jù)填充至全基因組序列數(shù)據(jù)。然后,基于隨機回歸測定日模型使用填充的序列數(shù)據(jù)對產(chǎn)奶性狀進行縱向全基因組關(guān)聯(lián)分析。縱向全基因組關(guān)聯(lián)分析共發(fā)現(xiàn)了與產(chǎn)奶量、乳脂率和乳蛋白率相關(guān)的130個QTL區(qū)域。
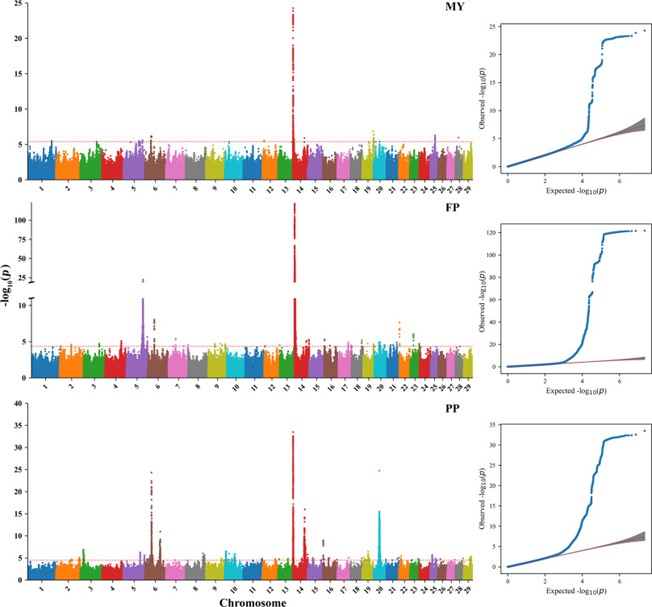
圖1. 產(chǎn)奶量、乳脂率和乳蛋白率縱向全基因組關(guān)聯(lián)分析的曼哈頓圖和QQ圖
為了進一步確定候選基因,該研究使用logP drop的方法估計每個QTL區(qū)域的95%置信區(qū)間。在這些置信區(qū)間中,共發(fā)現(xiàn)581個候選基因。此外,該研究重點關(guān)注僅包含1個基因或特別顯著top SNP的置信區(qū)間,在這樣的置信區(qū)間中發(fā)現(xiàn)了28個候選基因。其中大部分已在文獻中報道與奶牛產(chǎn)奶性狀有關(guān),如DGAT1、HSF1、MGST1、GHR、ABCG2、ADCK5和CSN1S1等。在未報道的基因中,CCSER1、CUX2、SNTB1、RGS7、OSR2和STK3等候選基因顯示出良好的潛力,值得進一步研究。
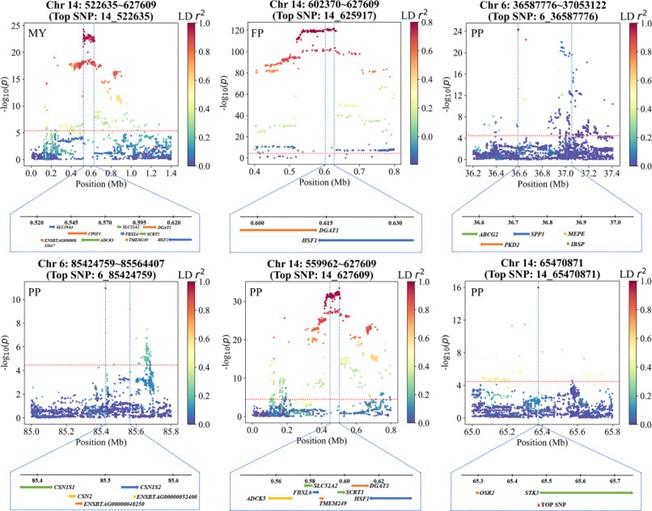
圖2. 包含特別顯著top SNP的QTL區(qū)域的95%置信區(qū)間
該研究不僅為產(chǎn)奶性狀的候選基因提供了新的見解,而且為使用序列數(shù)據(jù)基于隨機回歸測定日模型的縱向全基因組關(guān)聯(lián)分析提供了一般性的分析框架。
該研究得到了國家重點研發(fā)計劃、揚州大學(xué)畜牧學(xué)學(xué)科特區(qū)學(xué)科交叉課題、國家自然科學(xué)基金、山東省農(nóng)業(yè)良種工程、山東省自然科學(xué)基金和國家現(xiàn)代農(nóng)業(yè)產(chǎn)業(yè)技術(shù)體系的資助。
論文鏈接:https://doi.org/10.3168/jds.2022-22277
編 輯:萬 千
審 核:賈 波




