近日,動物科技學院張勤教授團隊在《Communications Biology》在線發表了題為“Improving multi-trait genomic prediction by incorporating local genetic correlations”的研究論文,該研究提出了三種整合局部遺傳相關信息的多性狀基因組預測方法用于提高基因組預測準確性。
基因組預測在推動人類精準醫療以及加速動植物遺傳改良方面具有巨大潛力。多性狀基因組預測模型的預測準確性往往要優于單性狀模型。然而,傳統的多性狀基因組預測模型(例如MTGBLUP)只考慮了性狀間的整體遺傳相關。隨著局部遺傳相關(LGC)估計方法的發展,現在可以分析特定基因組區域的性狀間遺傳相關,將局部遺傳相關信息納入多性狀基因組預測模型有望提高預測準確性。我們提出了三種方法(LGC-model-1、LGC-model-2和LGC-model-3)來解決這一問題,并使用模擬數據和人類、牛和豬三個真實數據集評估了新方法的性能。研究結果表明,局部遺傳相關在整個基因組中具有異質性。另外,與人類PRS模型(PRSice-2(C+T)、LDpred2和wMT-BLUP)和傳統的多性狀基因組預測模型(MTGBLUP)相比,三種方法可使預測準確性平均提高~12.76%。我們的研究結果凸顯了考慮局部遺傳相關信息對改進多性狀基因組預測的重要性。
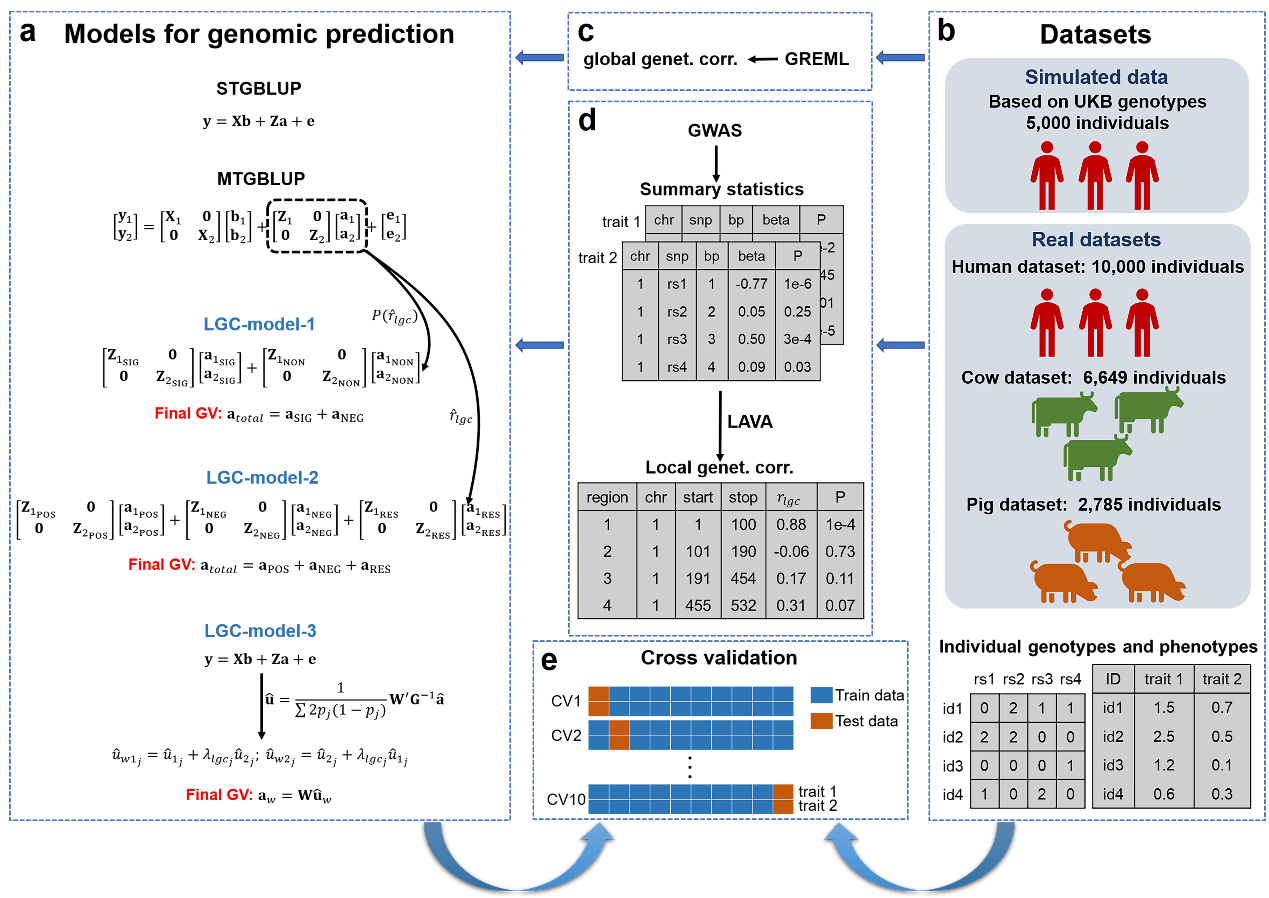
團隊博士后滕軍為該論文的第一作者,張勤教授和王丹教授為通訊作者。該研究得到國家重點研發計劃、生物育種重大項目、揚州大學畜牧學學科特區學科交叉課題和山東省農業良種工程等項目的資助。
論文鏈接:https://doi.org/10.1038/s42003-025-07721-9
編 輯:萬 千
審 核:賈 波









